前期回顾
PART1:自噬的概念及自度噬切入角度
PART2:自噬的“黄金标准” 及调控自噬流的方法
PART3
汉恒生物自噬领域的金牌研究工具
上面介绍了如何操纵自噬,但操纵自噬还必须要有观测手段来体现这种干预。下面介绍的荧光蛋白标记探针就是针对这种需求而开发,如LC3(LC3B)和p62定位标记的系列工具等,详述如下
①主流定位示踪工具- LC3
LC3是用来标记定位自噬小体到自噬溶酶体的动态变化,特别是利用mRFP-GFP-LC3差异化标记酸性和中性LC3阳性的囊泡,用颜色变化来指示自噬的不同阶段(由于GFP 荧光蛋白对酸性敏感,当自噬体与溶酶体融合后GFP 荧光发生淬灭,此时只能检测到红色荧光),可以在活细胞水平动态监测自噬流的强弱,满足定性和定量分析的要求。
GFP-LC3和RFP-LC3单标探针可以监测LC3蛋白参与自噬起始过程:自噬较少时,GFP-LC3和RFP-LC3融合蛋白主要弥散在胞浆中;自噬增加时,GFP-LC3和RFP-LC3融合蛋白转位至自噬体膜,在荧光显微镜下形成多个明亮的荧光斑点,一个斑点相当于一个自噬体,可以通过计数来评价自噬活性的高低。
我们可以在不同的药物处理时间点上采样进行静态分析,综合一起形成自噬流的动态分析;但RFP-LC3与mRFP-GFP-LC3的还可以对细胞进行活细胞成像,从而对自噬流进行动态监测。比如对同一个视野每隔5 min采集一次数据等。
对于单标探针(GFP-LC3或RFP-LC3)与双标探针(mRFP-GFP-LC3)的主要区别需要作进一步的说明:GFP-LC3和RFP-LC3单标探针对于监测自噬体的形成特别有用,但这两种单标探针不利于自噬流的整个过程进行动态检测,比如无法精确监测溶酶体是否参与,也无法示踪自噬体的成熟过程等。
因此,单标探针往往需要联合其他探针,比如溶酶体探针(Lamp1-RFP、Lamp1-GFP)一起使用,根据共定位的信息来更加精细的示踪自噬体的成熟过程。而mRFP-GFP-LC3串联双标记探针则可以解决这个问题,比如从自噬小体到自噬溶酶体的动态变化等(图4)。进一步的,若联合红外标记的溶酶体探针(LysoTracker® Deep Red ,Thermo,L12492)则可以进一步研究自噬小体/自噬溶酶体/溶酶体三者的动态变化。
需要注意的是:外源过表达的LC3如果过量,会导致LC3非特异性的自发聚集,为了区分自发聚集与自噬诱导的聚集,我们提供了不能参与自噬的LC3突变体(G120A)来作为对照。

图4,对于mRFP-GFP-LC3双标记探针标记的细胞,加入氯喹(chloroquine diphosphate,HeLa, 90 mM,处理24hr,通过中和溶酶体的酸性pH从而抑制自噬流)可以观察到红色荧光和绿色荧光同时增强;而加入蛋白酶抑制剂(如Leupeptin,HeLa, 200 mM,处理24hr,有效抑制溶酶体的酶活性,但不会改变溶酶体的pH)仅可以增加红色点状的强度,绿色荧光被酸性pH淬灭。
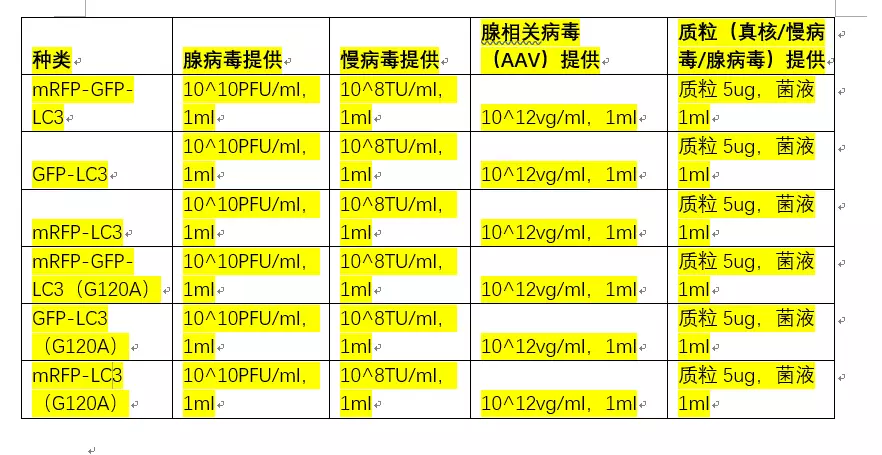
汉恒生物作为国内最先研究自噬的公司,有多种LC3标记的病毒,用户发表文章数百篇,包括cell等权威杂志。公司不仅有用于细胞实验的慢病毒和腺病毒以及质粒,还有用于动物实验的腺相关病毒,多种规格,多种荧光标记,列表如下:
② 主流定位示踪工具- p62
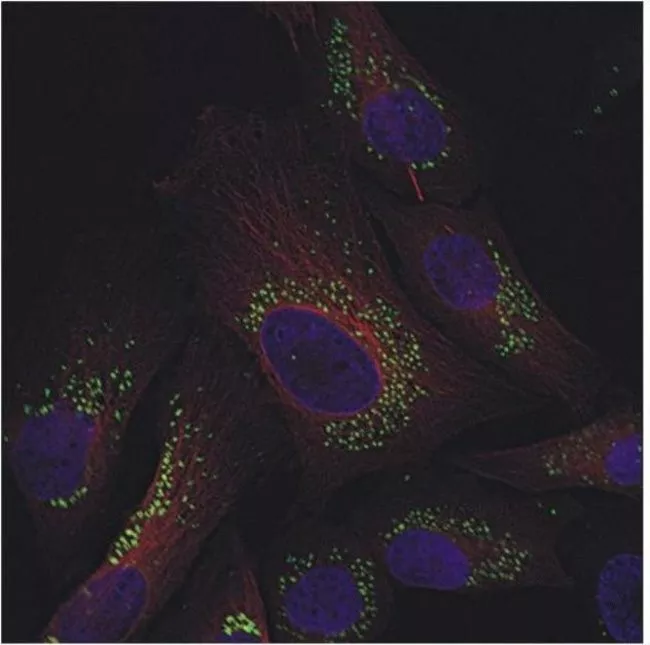
p62又称为 SQSTM1,是一种泛素结合蛋白,介导底物与LC3的结合,从而被选择性的转移到自噬机器中进行降解。P62通常被看作自噬降解蛋白质底物的一个标志性marker,其亚细胞的定位可以用来监测自噬的发生、蛋白底物的清除,以及自噬的抑制等过程。自噬被诱导时,p62定位在自噬小体并随后被递送到自噬溶酶体降解。相反,如果抑制自噬,则p62蛋白会积累在自噬小体(图5,图6)。
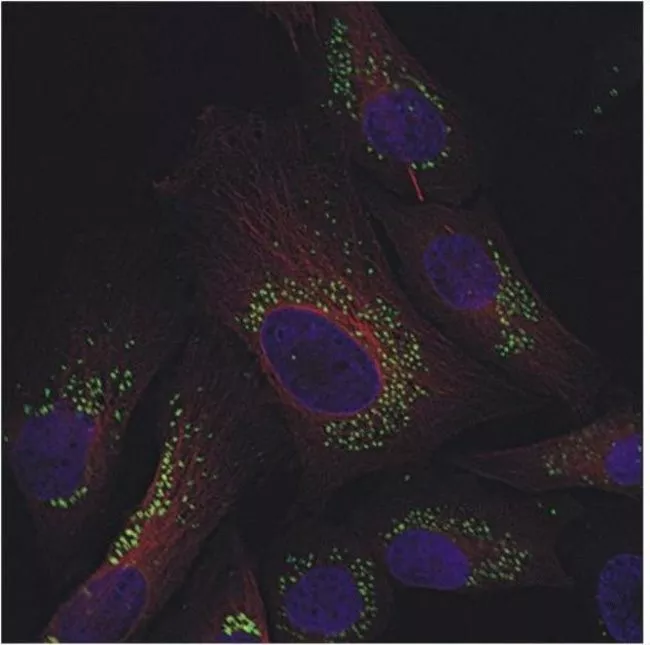
图5,A549细胞用GFP-p62和Tubulin-RFP同时标记。然后用60 mM 氯喹处理16hr。可以观察到明显的绿色点状自噬小体或自噬溶酶体沿着微管分布。
GFP-p62可以结合其他红色探针(如Tubulin-RFP、Lamp1-RFP、RFP-LC3等)联合使用。当自噬被诱导时,GFP-p62被招募到自噬小体(绿点)。此时如果加入氯喹抑制自噬流,则GFP-p62会累积在自噬小体和自噬溶酶体。
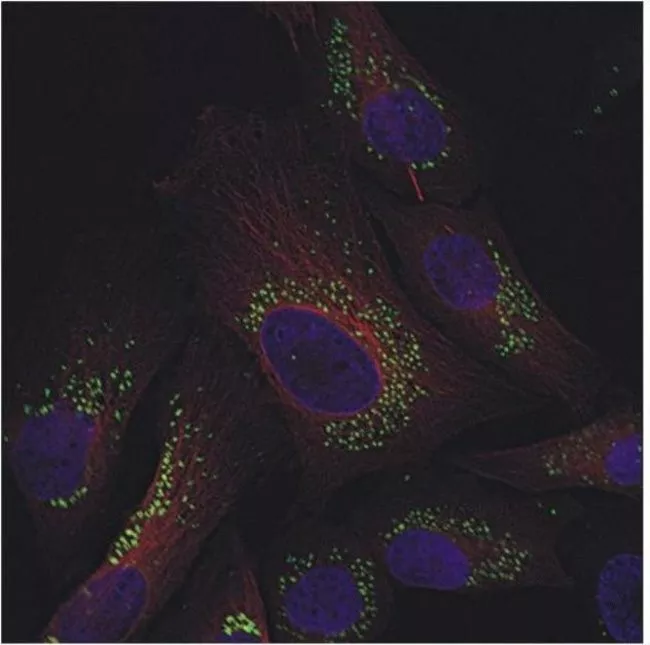
图6,U2OS用GFP-p62和LC3-RFP同时标记。然后用60 mM 氯喹处理16hr。可以观察到二者有着明显的共定位。
汉恒生物拥有常见物种人、大鼠、小鼠等的P62过表达载体,包括质粒、慢病毒、腺病毒、腺相关病毒现货等。
③ 其他定位工具-Keima探针
除了上面介绍的“金牌”工具之外,还有一些应用并不是那么广泛,但十分强大的自噬研究工具,如一种叫做Keima 的探针。Keima探针其实是一种pH敏感的荧光蛋白,它可以用来做经典的自噬(如LC3-Keima等);还可以用来研究选择性自噬,比如还可以改造定位在mito、Ribo、ER、Golgi、perioxsome等细胞器上来监视特殊细胞器的自噬。
图7示意的是用Ribo-Keima(核糖体定位的Keima)来研究核糖体相关的选择性自噬。线粒体定位的Keima探针在线粒体自噬的研究中应用非常广泛,具体信息可以参考“六、其他细胞器的选择性自噬研究”部分。
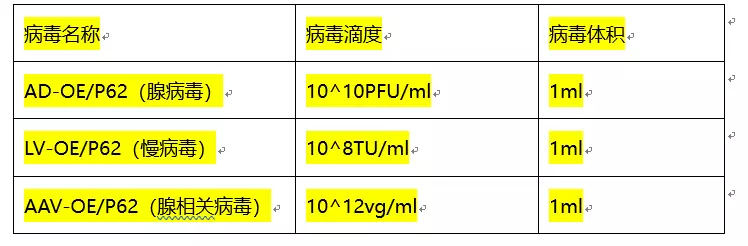
图7, Keima探针研究核糖体选择性自噬的基本原理。使用核糖体定位信号把Keima定位在核糖体上,然后研究核糖体自噬。由于Keima在不同的pH值下对应的激发光波长不同,在中性环境下只能被440 nm激发;在自噬溶酶体的酸性pH环境下,Keima只能被550 nm激发。其定量方法是用440 nm和550 nm两个不同波长的激光分别激发,然后用同一个发射光620 nm波长分别采集图像,然后用550 nm 激发对应的620 nm采集的图像荧光强度/ 440 激发对应的620 nm采集的图像荧光强这一比值度来指示pH值的变化:比值越大,pH值越低,反之比值越小,pH约偏于中性。以此来指示标记的细胞器所处的环境与位置。
今天的内容就是这些,下期我们将介绍:自噬研究过程中基因的操作。
我们下期见。
- 浏览 9015 次