LncRNA简介
LncRNA (Long noncoding RNAs, LncRNAs) 长链非编码RNA, 起初被认为是基因组转录的“噪音”,是RNA聚合酶II转录的副产物,不具有生物学功能。然而,近年来的研究表明,LncRNA参与了X染色体沉默(图A),基因组印记以及染色质修饰,转录激活(图B),转录干扰,核内运输等多种重要的调控过程,LncRNA的这些调控作用也开始引起人们广泛的关注。
哺乳动物基因组序列中4%~9%的序列产生的转录本是LncRNA(相应的蛋白编码RNA的比例是1%),虽然近年来关于LncRNA的研究进展迅猛,但是绝大部分LncRNA的功能仍不清楚,随着研究的推进,各类 LncRNA 的大量发现,LncRNA 的研究将是 RNA 基因组研究非常吸引人的一个方向,使人们逐渐认识到基因组存在人类知之甚少的“暗物质”。

图A:LncRNA参与X染色体的沉默; 图B:LncRNA参与基因的转录激活。
服务介绍
汉恒生物的LncRNA专家团队可以为您提供从LncRNA筛选、验证,LncRNA的功能探索以及更深层次的靶点寻找和表达调控机制探索等全方位的服务。
1. 差异性的LncRNA的筛选
A、生物信息学预测
根据客户提供的转录组信息、基因特征,通过与数据库(agilent_ncRNA、LncRNAdb、Gencode V13、H-invDB、UCSC_lincRNAsTranscripts等)比较,去除已知可能蛋白编码转录本、tRNA、rRNA、repeat等预测可能的LncRNA
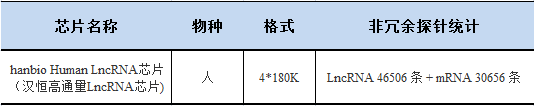
B、高通量的表达谱芯片
C、新一代的测序
具有现代的DNA测序技术的快速测序速度已经有助于达到测序完整的DNA序列。通过逆转录法,将LncRNA逆转录成cDNA,确定某一段序列,再通过RACE方法,确定已知序列的两端的序列。
2.生物信息学分析
根据高通量芯片数据,经过大型服务器处理,进行以下几项专业的生物信息学分析:
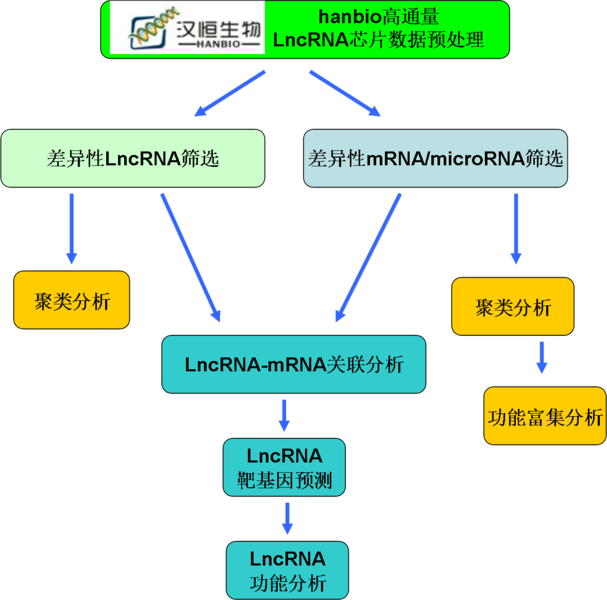
A、芯片数据预处理:对实验数据质量评估,预处理及均一化处理。
B、差异LncRNA和mRNA 的筛选:

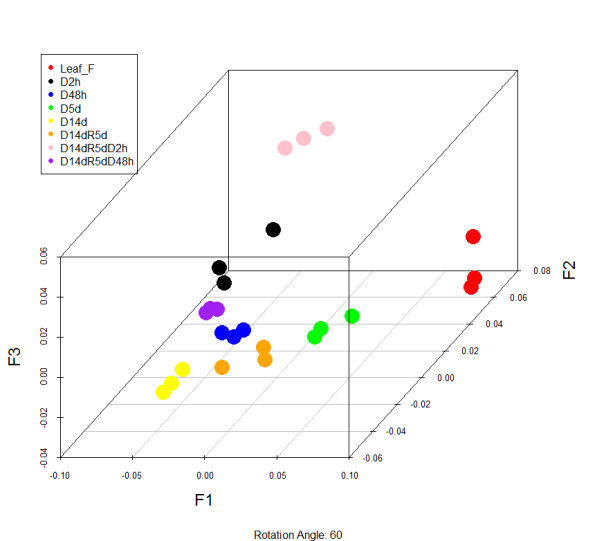
图:差异LncRNA和mRNA 的筛选
C、聚类分析:针对芯片结果进行样本及差异表达LncRNA和mRNA的聚类,寻找属于同一表达趋势的基因或样本。

图:聚类分析
D、功能富集分析:差异基因,应用数据库进行功能富集分析,挖掘具有统计学意义的差异表达基因的功能类别。


图:功能富集分析聚类分析
E、LncRNA-mRNA/microRNA相关性分析:根据设定的阈值筛选LncRNA与mRNA/microRNA关系对,构建LncRNA-mRNA共表达网络。
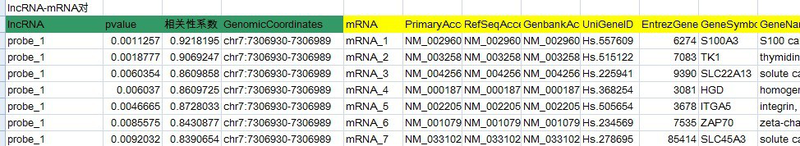
图:LncRNA-mRNA/microRNA相关性分析
F、LncRNA靶基因预测:基于LncRNA与mRNA/miRNA表达相关性以及LncRNA与mRNA/microRNA基因组位置近邻关系,得到LncRNA的潜在靶标基因。以及通过网络数据库miR CODE、Cat RAPID等进行预测LncRNA可能调控的miRNA和基因。(个性化定制)

图:miRcode数据库

图:CatRAPID数据库
G、LncRNA功能分析:对于已知功能的LncRNA,我们对LncRNA进行功能标注。对于未知功能的LncRNA,我们预测其功能,并进行功能富集分析。(个性化定制)
3、LncRNA的功能研究
A、LncRNA的表达变化-汉恒生物独有高灵敏度LncRNA检测技术(LncPrimerTM)
通过汉恒生物独有高灵敏度LncRNA检测引物库(LncPrimerTM),利用QPCR、FISH技术,在各种细胞、动物模型中,检测LncRNA在细胞和组织水平的分布和变化。
B、Gain of Function(体内和体外)
利用汉恒生物HanbioTM系列高效表达病毒载体(慢病毒、腺病毒等)全长过表达LncRNA
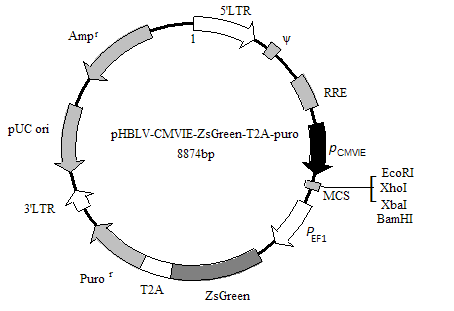
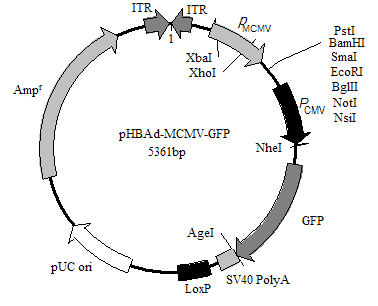
图:汉恒生物HanbioTM系列-高效过表达慢病毒和腺病毒载体
C、Loss of Function(体内和体外)
◆ 合成片段siRNA;
◆ 汉恒生物HanbioTM系列高效表达病毒载体(慢病毒、腺病毒等)表达shRNA;
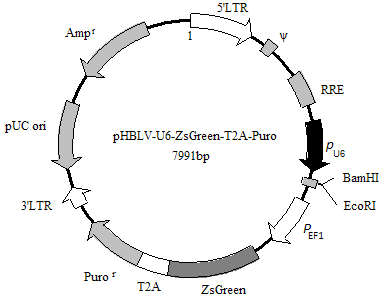
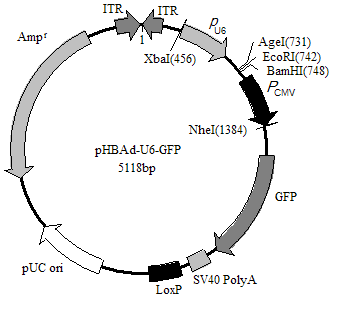
图:汉恒生物HanbioTM系列-高效下调慢病毒和腺病毒载体
◆ ASO(antisenseoligonucleotide,反义寡核苷酸),主要针对细胞核内的反义RNA起作用。
D、LncRNA沉默与定位分析(Combined knockdown and localization analysis of noncoding RNAs, c-KLAN)技术
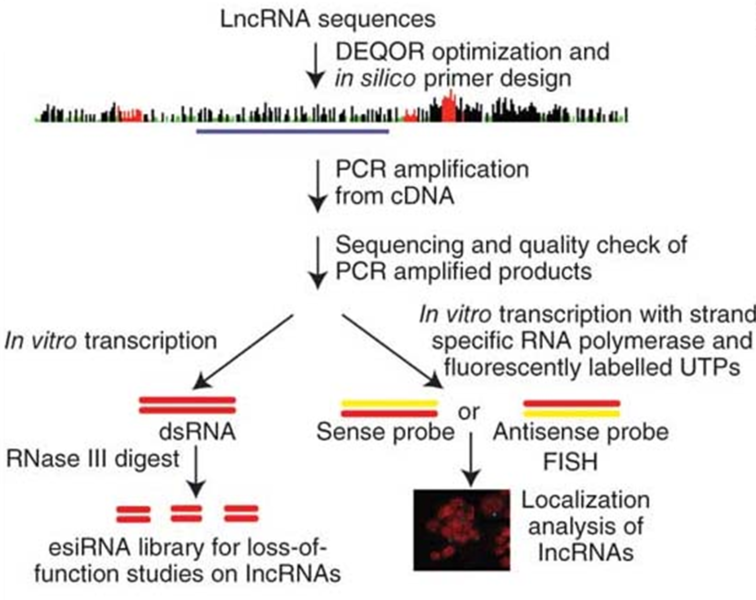
图:c-KLAN技术
4、LncRNA的调控机制研究
A、表观遗传研究-DNA甲基化
利用MSP、BSP技术,研究LncRNA对DNA甲基化的调控。
B、表观遗传研究-染色质重塑
利用ChIP技术,研究LncRNA对染色质重塑的调控。
C、转录调控研究
通过EMSA、RIP(下图所示)、ChIRP、Co-IP等技术,研究LncRNA与转录因子间的相互作用。
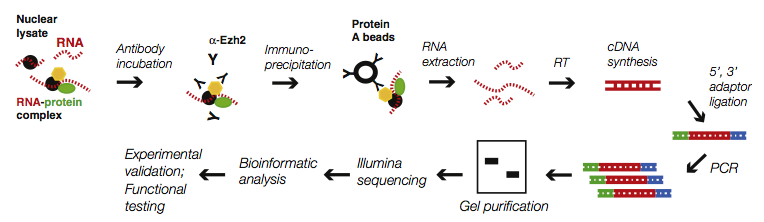
图:RIP技术流程
技术优势
1.汉恒生物拥有由若干LncRNA研究专家组成的专业LncRNA研究团队,技术水平卓越,专注于LncRNA整体服务,可为您提供一体化的LncRNA系统研究方案;
2.汉恒生物已成功完成许多LncRNA项目,拥有成熟的LncRNA相关经验;
3.如需要详细的LncRNA课题/实验指导,欢迎登陆汉恒官网www.hanbio.net与汉恒生物的LncRNA专家交流探讨更多细胞功能学研究领域。直击以下链接通过QQ与我们的技术工程师一对一交流!
http://wpa.b.qq.com/cgi/wpa.php?ln=1&key=XzgwMDA1Mjg2M18xMjcwNzFfODAwMDUyODYzXzJf
参考文献
[1] Beltran M, Puig I, Peña C, et al. (March 2008). "A natural antisense transcript regulates Zeb2/Sip1 gene expression during Snail1-induced epithelial-mesenchymal transition". Genes & Development 22 (6): 756–69. doi:10.1101/gad.455708. PMC 2275429. PMID 18347095. [2] Bentwich I, Avniel A, Karov Y, et al. (July 2005). "Identification of hundreds of conserved and nonconserved human microRNAs". Nature Genetics 37 (7): 766–70. doi:10.1038/ng1590. PMID 15965474. [3] Birney E, Stamatoyannopoulos JA, Dutta A, et al. (June 2007). "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project". Nature 447 (7146): 799–816. Bibcode:2007Natur.447..799B. doi:10.1038/nature05874. PMC 2212820. PMID 17571346. [4] Braidotti G, Baubec T, Pauler F, et al. (2004). "The Air noncoding RNA: an imprinted cis-silencing transcript". Cold Spring Harbor Symposia on Quantitative Biology 69: 55–66. doi:10.1101/sqb.2004.69.55. PMID 16117633. [5] Brosius J (May 2005). "Waste not, want not--transcript excess in multicellular eukaryotes". Trends in Genetics 21 (5): 287–8. doi:10.1016/j.tig.2005.02.014. PMID 15851065. [6] Calin GA, Liu CG, Ferracin M, et al. (September 2007). "Ultraconserved regions encoding ncRNAs are altered in human leukemias and carcinomas". Cancer Cell 12 (3): 215–29. doi:10.1016/j.ccr.2007.07.027. PMID 17785203. [7] Costanzi C, Pehrson JR (June 1998). "Histone macroH2A1 is concentrated in the inactive X chromosome of female mammals". Nature 393 (6685): 599–601. Bibcode:1998Natur.393..599C. doi:10.1038/31275. PMID 9634239. [8] Derrien, T.; Johnson, R.; Bussotti, G.; Tanzer, A.; Djebali, S.; Tilgner, H.; Guernec, G.; Martin, D.; Merkel, A.; Knowles, D. G.; Lagarde, J.; Veeravalli, L.; Ruan, X.; Ruan, Y.; Lassmann, T.; Carninci, P.; Brown, J. B.; Lipovich, L.; Gonzalez, J. M.; Thomas, M.; Davis, C. A.; Shiekhattar, R.; Gingeras, T. R.; Hubbard, T. J.; Notredame, C.; Harrow, J.; Guigó, R. (2012). "The GENCODE v7 catalog of human long noncoding RNAs: Analysis of their gene structure, evolution, and expression". Genome Research 22 (9): 1775–1789. doi:10.1101/gr.132159.111. [9] Faghihi MA, Modarresi F, Khalil AM, et al. (July 2008). "Expression of a noncoding RNA is elevated in Alzheimer's disease and drives rapid feed-forward regulation of beta-secretase". Nature Medicine 14 (7): 723–30. doi:10.1038/nm1784. PMC 2826895. PMID 18587408. [10] Feng J, Bi C, Clark BS, Mady R, Shah P, Kohtz JD (June 2006). "The Evf-2 noncoding RNA is transcribed from the Dlx-5/6 ultraconserved region and functions as a Dlx-2 transcriptional coactivator". Genes & Development 20 (11): 1470–84. doi:10.1101/gad.1416106. PMC 1475760. PMID 16705037. [11] Hesman Saey, Tina (17 December 2011). "Missing Lincs". Science News 180 (13): 22–25.Ishii N, Ozaki K, Sato H, et al. (2006). "Identification of a novel non-coding RNA, MIAT, that confers risk of myocardial infarction". Journal of Human Genetics 51 (12): 1087–99. doi:10.1007/s10038-006-0070-9. PMID 17066261. [12] Lin R, Maeda S, Liu C, Karin M, Edgington TS (February 2007). "A large noncoding RNA is a marker for murine hepatocellular carcinomas and a spectrum of human carcinomas". Oncogene 26 (6): 851–8. doi:10.1038/sj.onc.1209846. PMID 16878148. [13] Mohammad F, Pandey RR, Nagano T, et al. (June 2008). "Kcnq1ot1/Lit1 noncoding RNA mediates transcriptional silencing by targeting to the perinucleolar region". Molecular and Cellular Biology 28 (11): 3713–28. doi:10.1128/MCB.02263-07. PMC 2423283. PMID 18299392. [14] Pang KC, Frith MC, Mattick JS (January 2006). "Rapid evolution of noncoding RNAs: lack of conservation does not mean lack of function". Trends in Genetics 22 (1): 1–5. doi:10.1016/j.tig.2005.10.003. PMID 16290135. [15] Reinius, B.; Shi, C.; Hengshuo, L.; Sandhu, K.; Radomska, K. J.; Rosen, G. D.; Lu, L.; Kullander, K.; Williams, R. W.; Jazin, E. (2010). "Female-biased expression of long non-coding RNAs in domains that escape X-inactivation in mouse". BMC Genomics 11: 614. doi:10.1186/1471-2164-11-614. PMC 3091755. PMID 21047393.. [16] Zearfoss NR, Chan AP, Kloc M, Allen LH, Etkin LD (April 2003). "Identification of new Xlsirt family members in the Xenopus laevis oocyte". Mechanisms of Development 120 (4): 503–9. doi:10.1016/S0925-4773(02)00459-8. PMID 12676327.
- 浏览 13243 次